Valutazione di un classificatore
Considerata l'impossibilità di costruire un classificatore universale migliore in assoluto, dovendo quindi valutare per ogni problema quale tecnica risulti migliore, sorge il problema di poter valutare la bontà di un classificatore applicato a un dato problema di classificazione. Si osservi che per la valutazione di un classificatore l'unico strumento utilizzabile è il classificatore stesso ed il dataset di addestramento.
Contents
Valutazione di un singolo test
Per la valutazione delle performance di un classificatore devono essere seguite le fasi di addestramento e test su dataset differenti, per questo motivo il dataset di addestramento viene suddiviso in due parti, una delle quali viene utilizzata per l'addestramento e l'altra per il test.
La valutazione della bontà di un classificatore avviene calcolando alcuni coefficienti tra cui:
Accuratezza: Percentuale di classificazioni corrette. Precision: Percentuale di positivi trovati. Recall: Percentuale di documenti trovati.
Per il calcolo di queste misure spesso si ricorre all'utilizzo di:
TP: Numero di veri positivi (classificati true erano true). TN: Numero di veri negativi (classificati false erano false). FP: Numero di falsi positivi (classificati true anche se false). FN: Numero di falsi negativi (classificati false anche se true).
da cui:
accuracy = \frac{TP+TN}{TP+TN+FP+FN}
precision = \frac{TP}{TP+FP}
recall = \frac{TP}{TP+FN}% Carico il dataset: load mnist_01r2; % Preparo i dati di train: samples = double([train0,train1]); classification = [false(1,size(train0,2)),true(1,size(train1,2))]; % Addestro un classificatore: net = OIPCAClearn(samples,classification); % Preparo il dataset di test: testSamples = double([test0,test1]); testClassification = [false(1,size(test0,2)),true(1,size(test1,2))]; % Eseguo il test: sclass = OIPCACclassify(net,testSamples); % Calcolo l'accuratezza: acc = sum(sclass==testClassification)/numel(sclass); % Calcolo TP, TN, FP, FN: TP = sclass & testClassification; TN = not(sclass) & not(testClassification); FP = sclass & not(testClassification); FN = not(sclass) & testClassification; % Precision e recall: precision = TP/(TP+FP); recall = TP/(TP+FN); % Vediamo i risultati: fprintf('accuracy: %2.2f%%\nprecision: %2.2f%%\nrecall: %2.2f%%\n',acc*100,precision*100,recall*100);
accuracy: 99.81% precision: 99.91% recall: 99.74%
K-Folding
Una valutazione più accurata di un classificatore può essere ottenuta iterando la procedura seguente sull'intero dataset operando come segue:
1) fissato un valore intero K>1, il dataset viene partizionato; 2) ad ogni iterazione K-1 parti vengono usate per l'addestramento e una per il test; 3) le misure di bontà del classificatore vengono mediate.
Se si utilizza K=N numero totale di esempi si ottiene leave-one-out, ad ogni iterazione tutto il dataset tranne un elemento viene utilizzato per l'addestramento e un solo esempio viene usato per il test, l'insieme finale di classificazioni viene usato per il calcolo delle misure di bontà del classificatore.
Altre misure spesso utilizzate sono:
Specificity: Abilità nell'identificare i negativi.
specificity = \frac{TN}{TN+FP}
Sensitivity: Abilità nell'identificare i positivi.
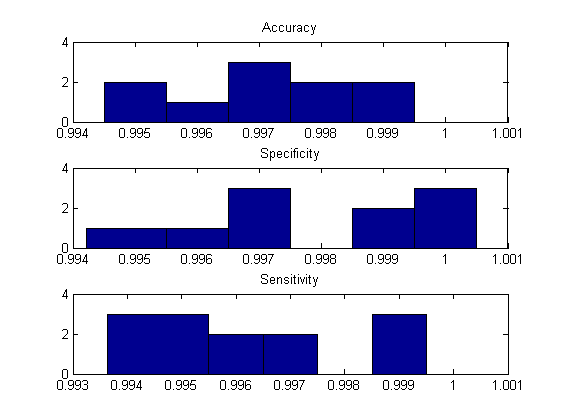
specificity = \frac{TP}{TP+FN}% Preparo i dati: samples = [samples,testSamples]; classification = [classification,testClassification]; % Esecuzione del K-Folding: [accuracies,specificity,sensitivity] = ... KFolding(samples,classification,'OIPCAC',{},10); % Vediamo i risultati: figure; Hx = 0.995:0.001:1; subplot(311); hist(accuracies,Hx); title('Accuracy'); subplot(312); hist(specificity,Hx); title('Specificity'); subplot(313); hist(sensitivity,Hx); title('Sensitivity');